
В этом руководстве вы узнаете, как использовать функции обработки изображений MatLab, чтобы сделать МРТ-сканирование мозга с опухолью и выделить изображение, чтобы показать только опухоль, а также дать некоторые анатомические подробности о ней. Перед запуском рекомендуется обновить MatLab, а также получить некоторые предварительные базовые знания в области программирования или обработки изображений.
Шаг 1: поиск подходящих изображений МРТ
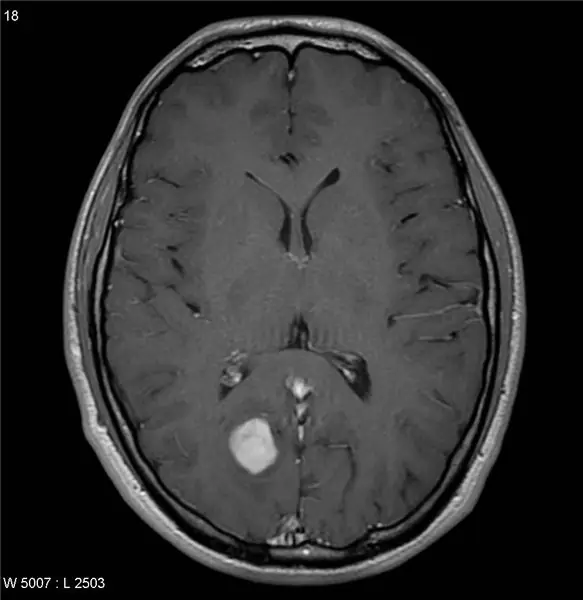
Конечно, любой может сделать некачественный снимок с изображений Google… но вы лучше этого! Хотя иногда бывает довольно сложно найти такие виды сканирования, важно убедиться, что вы используете правильные изображения без каких-либо водяных знаков или дополнительного шума из-за плохой обработки изображений со сторонних веб-сайтов. Есть много онлайн-баз данных, которые публикуют изображения для исследовательских целей, которые вы можете использовать. Некоторые базы данных включают Aylward и OpenfMRI. Вот ссылка для начала: https://www.aylward.org/notes/open-access-medical-…. Четкое и точное изображение МРТ - это первый шаг к тому, чтобы стать профессионалом в области обработки изображений.
Шаг 2: преобразование в двоичное изображение
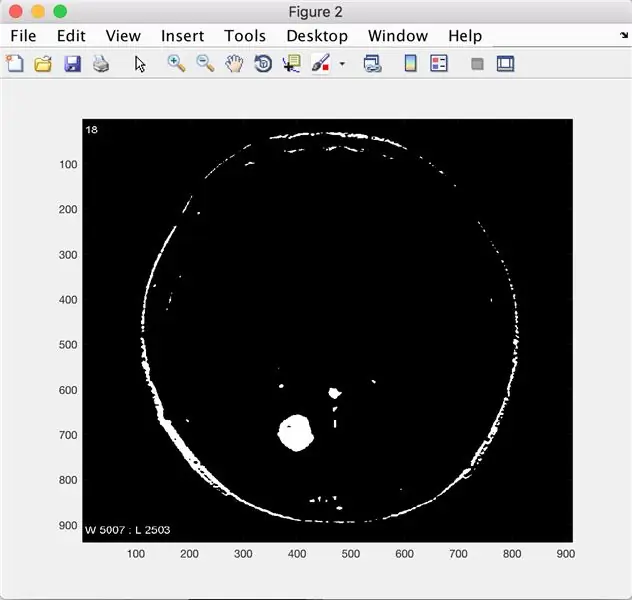
Чтобы правильно обработать ваше изображение, вы хотите сначала преобразовать его в двоичное изображение. Бинарное изображение - это просто изображение, состоящее из единиц и нулей, соответствующих черным или белым пикселям. Для вашего изображения мы собираемся установить пороговое значение для нашего изображения, чтобы оно принимало значения пикселей, превышающие установленный порог (возможно, вам придется немного поиграть с числом. Может быть, начните со 150) и преобразует их в единицы. Таким образом, мы можем немедленно избавиться от любых участков изображения, которые слишком темны, чтобы быть опухолью.
В Matlab мы можем сделать это, введя код:
I = imread ('имя_файла_образа');
фигура 2); thresha = I> 150 и I <260;
imagesc (thresha); палитра (серый);
Это отобразит двоичное изображение, которое удаляет слишком тусклые или слишком яркие пиксели, в этом случае значения пикселей от 150 до 260 будут установлены на 1, а другие значения - на 0.
Шаг 3. Поиск пороговых значений (необязательно)
Хотя это не совсем необходимо, мы чувствовали, что автоматизация порогового значения добавит немного большей точности нашему коду, а также позволит проводить динамический анализ большего количества изображений. Эта автоматическая пороговая функция основывалась на анализе интенсивности пикселей в каждом изображении для получения разброса значений пикселей. В MatLab это можно сделать, просто используя серию операторов if / elseif, вложенных в два цикла for, чтобы подсчитать, сколько раз встречается каждая запись пикселя. В приведенном ниже коде показан фрагмент этого процесса.
[iRow, iCol] = размер (ImageFile);
для iLoop = 1: iRow для iLoop0 = 1: iCol
если (I (iLoop, iLoop0)> = 250)
iPixelList (25) = iPixelList (25) + 1;
elseif (I (iLoop, iLoop0)> = 240)
iPixelList (24) = iPixelList (24) + 1;
Из последующих группировок этих значений интенсивности в ячейки с шириной 10 пикселей (как показано выше), подсчеты пикселей, которые были слишком малы по значению, игнорировались (1), самая большая последовательная группа этих значений была изолирована с помощью цикла for (2), индекс минимального и максимального значений общего количества записей пикселей был затем удален (3), и было вычислено среднее значение для оставшихся значений количества записей пикселей (4). Краткий фрагмент этого алгоритма показан ниже.
(1)
переключатель iLoop case 1
iFilter = 6000; % установить значение количества пикселей, которое следует игнорировать
чехол 2
iFilter = 5000;
чехол 3
iFilter = 4000;
конец
% цикл для удаления ненужных пикселей
для iLoop = 2: длина (iPixelList)
if (iCopyPixelList (iLoop) <iFilter)% проверить, не является ли нежелательный пиксель
iCopyPixelList (iLoop) = 0; % удалить значение пикселя из списка
конец
конец
(2)
iMax = 0; % цикл, чтобы найти самую большую группу пикселей
для iLoop = 1: длина (iPixelList)
если (iPixelGroup (iLoop)> iMax)
iMax = iPixelGroup (iLoop);
iBiggestPixelGroupIndex = iLoop;
конец
конец;
if (iBiggestPixelGroupIndex == 0)% проверяет, найдена ли самая большая группа пикселей
iThreshold = 0; % не присвоить порог
вернуть% ничего не найдено, затем выйти из функции
конец
(3)
iPixelList (iMinIndex) = 0;
iPixelList (iMaxIndex) = 0;
ОстающийсяПиксельГруппа = iPixelGroup (iBiggestPixelGroupIndex) - 2;
(4)
iAvg = iSum / unknownPixelGroup; % вычислить среднее значение самой большой группы пикселей
Затем функция использует три метода для определения наилучшего порогового значения. Первый метод использует значение интенсивности пикселей, наиболее близкое к индексу вычисленного среднего значения, в качестве порога, второй метод использует индекс минимального значения общего количества для сгруппированных записей пикселей в качестве порога, а третий метод использует индекс максимального значения общего количества записей сгруппированных пикселей в качестве порогового значения. Функция возвращает пороговое значение, которое дает наибольшее количество пикселей, чтобы избежать обрезки слишком большой части исходного изображения. Это делается следующим образом:
% найти средний / минимальный / максимальный порог, который возвращает наибольшее количество пикселей, если (iAvgThreshold> 0 && iAvgPixelCtr> iMinPixelCtr && iAvgPixelCtr> iMaxPixelCtr)
iThreshold = iAvgThreshold; % устанавливает окончательный порог
elseif (iMinThreshold> 0 && iMinPixelCtr> iAvgPixelCtr && iMinPixelCtr> iMaxPixelCtr)
iThreshold = iMinThreshold * 10; % устанавливает конечный порог
elseif (iMaxThreshold> 0)
iThreshold = iMaxThreshold * 10; % устанавливает конечный порог
конец
Теперь вы успешно разработали функцию для динамического определения подходящего порогового значения без использования метода проб и ошибок для преобразования вашего МРТ в двоичное изображение.
Шаг 4. Отфильтруйте шум на изображении
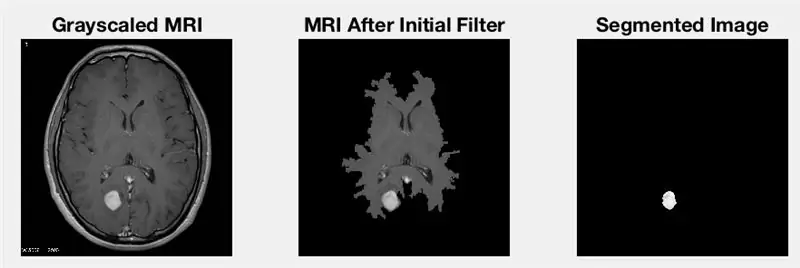
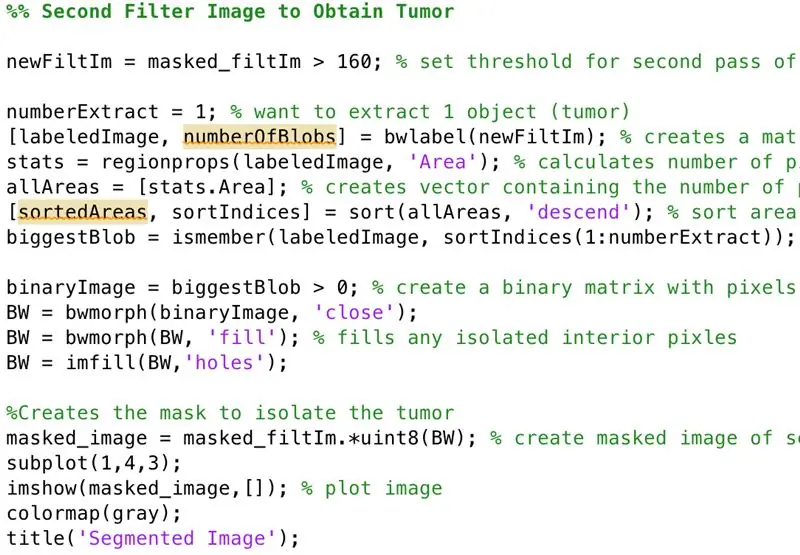
Теперь мы хотим избавиться от всех лишних деталей, таких как контур черепа или некоторых других пятен, которые все еще присутствуют на изображении. Существует множество различных методов фильтрации этих дополнительных разделов, которые вы можете использовать, и рекомендуется использовать несколько для получения наилучших результатов (дополнительная информация здесь: https://www.mathworks.com/help/ images /linear-filtering.html.)
Нашим первым шагом было выполнение ряда морфологических функций в коде (например, filterImage = bwmorph (filterImage, 'close')), чтобы выполнить очень простую фильтрацию и избавиться от некоторых простых фильтров на изображении. После этого мы использовали алгоритм, чтобы найти все капли на отфильтрованном изображении и отсортировать их по площади. Затем мы проверили, какие капли в голове имеют достаточно большую площадь, чтобы быть опухолью. Затем мы удалили все капли, которые были слишком маленькими, чтобы быть опухолью, и в итоге остались с последней опухолью. (См. Второе изображение выше для примера кода.) Наконец, мы создали замаскированное изображение, чтобы дополнительно изолировать предполагаемую интересующую область, увеличивая контраст между окружающими структурами и областью опухоли на МРТ-сканировании.
Шаг 5: выделение опухоли
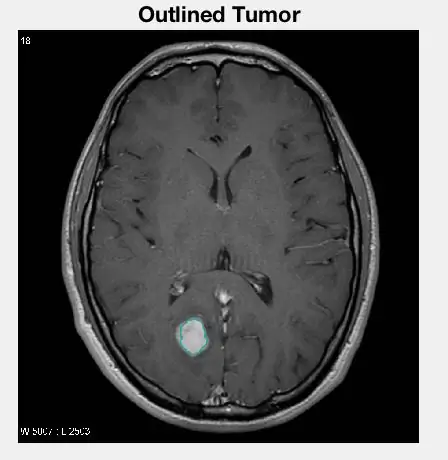
Наши последние операции, выполненные с изображением, позволили нам наложить цветную форму вокруг опухоли на исходном изображении, чтобы выделить ее для зрителя. Для выполнения этого шага мы использовали код:
BWoutline = bwperim (BW); % найти периметр опухоли на двоичном изображении SE3 = strel ('квадрат', 3); % использовать элемент структурирования для увеличения толщины контура на 3 пикселя в ширину
outlineImg = II;
ThickOutlines = imdilate (BWoutline, SE3); % увеличить толщину контура
OutlineImg (ThickOutlines) = 50; % установить цвет контура
подзаговор (1, 4, 4);
imshow (outlineImg); % отобразить очерченную опухоль на исходной МРТ
title («Обрисованная опухоль»);
Для этого мы сначала использовали другую морфологическую функцию, чтобы найти внешний периметр изолированной опухоли. Когда мы достигли периметра, мы установили для соответствующих пикселей исходного изображения значение 50, чтобы изменить их цвет и сделать их ярче. Наконец, после этого мы отобразили изображение на подзаголовке с другими желаемыми изображениями, полученными ранее.
Шаг 6: Обработка изображения и оценка анатомической информации
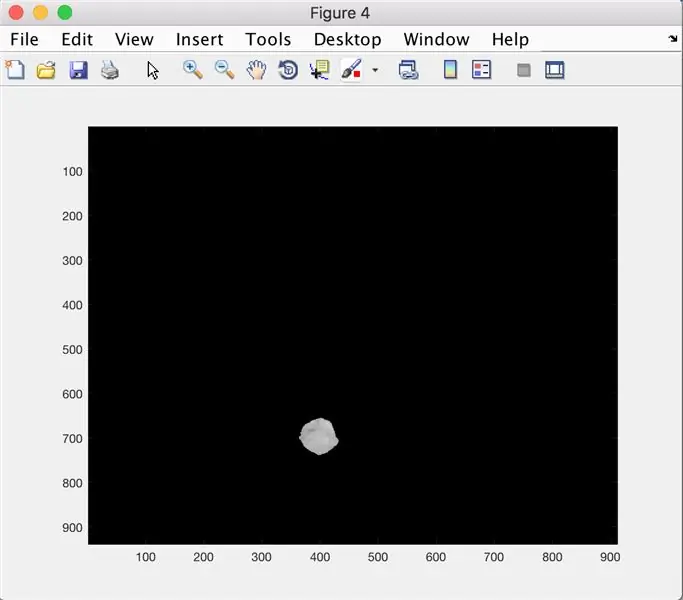

Теперь, когда у нас есть последняя опухоль, мы можем выполнить несколько функций с изображением, чтобы дать несколько более конкретных характеристик. В нашей программе мы хотели рассчитать размер, площадь и расположение опухоли по отношению к общему изображению. Для этого мы смогли использовать некоторые встроенные функции из MatLab, чтобы сделать этот процесс очень простым. Функция regionprops из Matlab позволяет нам вычислить большую часть желаемой информации с помощью всего нескольких строк кода (дополнительная информация здесь: https://www.mathworks.com/help/images/ref/regionp…). расположение и размер относительно изображения и сохранить его в текстовый файл, чтобы сохранить информацию, мы использовали этот код:
% Сохранение результатов в виде текстового файла FileName = 'MRI Brain Tumor Results';
fid = fopen (имя файла, 'вес'); % открывает файл
stats2 = regionprops (BW, 'Центроид', 'Площадь');
center = stats2. Centroid;
[строки, столбцы] = найти (BW);
Hrow = max (ряды); Lrow = min (ряды); rowBounds = [Хроу, Лроу];
Hcol = max (столбцы); Lcol = min (столбцы); colbounds = [Hcol, Lcol];
После использования параметра regionprops для определения местоположения центра опухоли и количества пикселей, которые он заполняет, мы захотели найти область, которую охватывает опухоль. Для этого мы нашли все строки и столбцы, в которых были единицы, затем провели поиск самой высокой и самой низкой строки и столбца, чтобы получить общее пространство, охватываемое опухолью. Чтобы вывести всю эту информацию в командное окно, мы использовали:
fprintf (fid, 'Центр опухоли расположен в координатах (% 0.f,% 0.f) n', center (1), center (2)); fprintf (fid, 'Опухоль опухоли простирается от От% 0.d до% 0.d по оси X и% 0.d и% 0.d по оси Y. / N ', colbounds (2), colbounds (1), rowBounds (2), rowBounds (1));
fprintf (fid, 'Опухоль занимает площадь% 0.d пикселей. / n', stats2. Area)
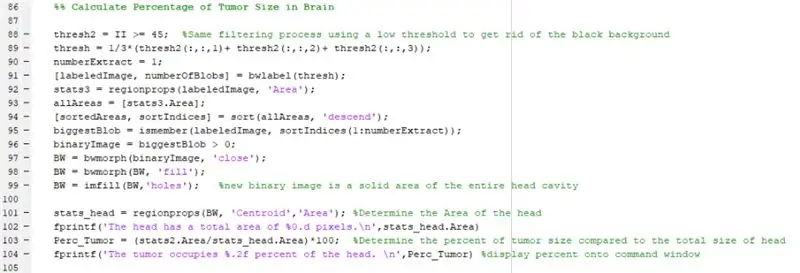
Если вы действительно хотите сделать все возможное, вы можете добавить несколько дополнительных строк кода, чтобы сообщить вам, какой процент мозга заполняет ваша опухоль. Следующий код ниже можно использовать в качестве ориентира, который поможет вам двигаться дальше:
thresh2 = II> = 45; % новый порог для избавления от черного фона thresh = 1/3 * (thresh2 (:,:, 1) + thresh2 (:,:, 2) + thresh2 (:,:, 3));
numberExtract = 1;
[labeleledImage, numberOfBlobs] = bwlabel (порог);
stats3 = regionprops (помеченное Изображение, 'Площадь');
allAreas = [stats3. Area];
[sortedAreas, sortIndices] = сортировать (allAreas, 'по убыванию');
самый большойBlob = ismember (labeleledImage, sortIndices (1: numberExtract)); % отфильтровать все, кроме головы в целом
binaryImage = BigBlob> 0;
BW = bwmorph (binaryImage, «закрыть»);
BW = bwmorph (BW, 'заполнить');
BW = imfill (BW, «дыры»);
Perc_Tumor = (stats2. Area / stats_head. Area) * 100;
Чтобы сравнить опухоль с головой в целом, мы сначала воссоздали другое двоичное изображение, но на этот раз с очень низким порогом, чтобы оно преобразовало всю структуру головы в двоичное изображение. После этого мы нашли самую большую каплю на изображении и удалили любой другой фоновый шум (потому что мы знаем, что иногда вам просто нужно использовать изображение с водяными знаками…), чтобы изолировать голову. Затем мы можем найти всю область головы, которую затем используем площадь опухоли, чтобы просто вычислить процент.
дополнительная информация о fprintf здесь: (https://www.mathworks.com/help/matlab/ref/fprintf.html)
Шаг 7: Заключение: конечный продукт и дальнейшие цели, которые могут быть достигнуты
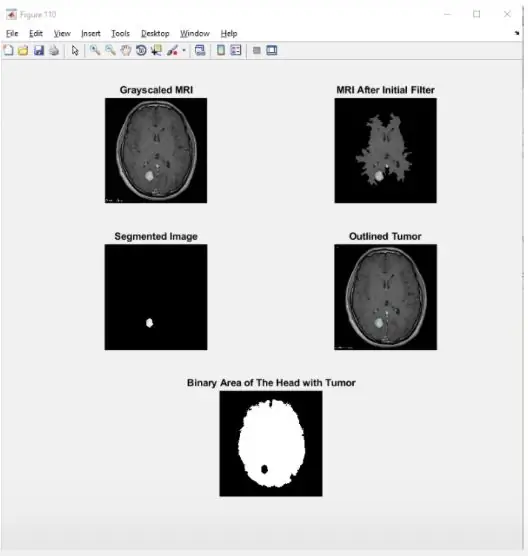
Поздравляю! Теперь вы закончили свой первый (возможно) проект MatLab по обработке изображений! Данные, полученные с помощью этого кода, являются отправной точкой в отношении диагностики опухолей головного мозга. Хотя нам удалось получить большую часть информации об опухоли, касающейся ее физического присутствия в головном мозге, важным шагом является фактическая классификация опухоли как доброкачественной или злокачественной. Мы можем сохранить это для следующего проекта! А пока вы можете похвастаться перед друзьями, какой удивительный и крутой гаджет вы только что построили на своем компьютере, не используя ничего, кроме хитрости и острого ума, а затем наблюдайте, как они постепенно перестают писать вам текстовые сообщения и находят новых и более интересных друзей. вместо. Но эй, это просто означает больше свободного времени, чтобы пойти и применить эти навыки в другом месте! Так что попробуйте что-нибудь еще с тем, что вы только что узнали. Есть много разных проектов, которые вы можете делать, и вы можете обрабатывать практически любое изображение, которое найдете, так что получайте удовольствие!
